Introdução ao Scikit-learn - Parte 3: avaliando a qualidade do modelo via cross-validation
Introdução
Esse é o terceiro post da série de tutorias sobre a Scikit-learn. Os dois primeiros posts podem ser acessados nos seguintes links:
Neste post, vamos utilizar o modelo criado no projeto passado para criarmos pipelines mais adequados para a avaliação de desempenho do mesmo. Como sabemos, algoritmos de machine learning aprendem a partir do dados. Além disso, muitos são estocásticos. Isso faz com que a avaliação desses modelos se torne extremamente importante para garantir que eles performem bem quando colocados em produção, ou seja, quando expostos a dados nunca antes visto. Vamos abordar duas maneiras comuns de avaliar modelos de ML: multiplas avaliações aleatórias e cross-validation.
Dica: antes de prosseguir, talvez você queira dar uma olhada no nosso post sobre os principais conceitos relacionados a Avaliação de modelos, cross-validation e data leakage.
Lembrando que todos os trechos de códigos utilizados neste post estão disponíveis no repositório do Github dessa série de posts.
Executando o mesmo pipeline multiplas vezes
Uma maneira comum de se avaliar o desempenho de um modelo treinado a partir de um conjunto de dados é executar o pipeline de treinamento e teste multiplas vezes e calcular a média e desvio padrão da métrica de desempenho escolhida. Para executar essa estratégia, vamos reutilizar o modelo e o conjunto de dados utilizado na parte 2:
from sklearn.datasets import load_wine
from sklearn.preprocessing import MinMaxScaler
from sklearn.neighbors import KNeighborsClassifier
from sklearn.pipeline import Pipeline
wine_dataset = load_wine()
X = wine_dataset['data']
y = wine_dataset['target']
nome_das_classes = wine_dataset.target_names
knn_pipeline = Pipeline(steps=[
("normalizacao", MinMaxScaler()),
("KNN", KNeighborsClassifier(n_neighbors=3))
])
Até aqui, nada novo. Apenas pegamos o modelo que utilizamos na parte 2. Agora, o que vamos fazer é o seguinte: particionar o dataset em treino e teste, treinar o modelo e calcular a acurácia atingida na partição de teste. Porém, vamos fazer isso N vezes:
from sklearn.model_selection import train_test_split
import numpy as np
N = 10
acuracias = list()
for i in range(N):
X_treino, X_teste, y_treino, y_teste = train_test_split(X, y, test_size=0.3, shuffle=True)
knn_pipeline.fit(X_treino, y_treino)
ac_i = knn_pipeline.score(X_teste, y_teste)
acuracias.append(ac_i)
print("- Acurácia:")
print(f"Media: {round(np.mean(acuracias) * 100, 2)}%")
print(f"Desvio padrão: {round(np.std(acuracias) * 100, 2)}%")
Neste exemplo, definimos N = 10. Dessa forma, o modelo seria treinado 10 vezes. Em cada iteração as partições de treino e teste são geradas de maneira aleatória. Observe que o parâmetro shuffle = True e não utilizamos nenhuma random_state. Além disso, separamos 30% para teste e 70% para treino. Na sequência, calculamos a acurácia e guardamos em um array. Desse array, extraímos a média e desvio padrão da métrica.
Obviamente, essa não é a melhor estratégia para avaliarmos nosso modelo. Existem vários problemas relacionados a vazamento de dados, multiplas execuções do modelo, que dependendo do tamanho do conjunto de dados e do modelo pode tomar muito tempo para executar, etc. Todavia, essa opção é melhor do que executar o modelo apenas uma vez e calcular a métrica de desempenho. Para entender melhor os problemas, dê uma olhada no post Avaliação de modelos, cross-validation e data leakage.
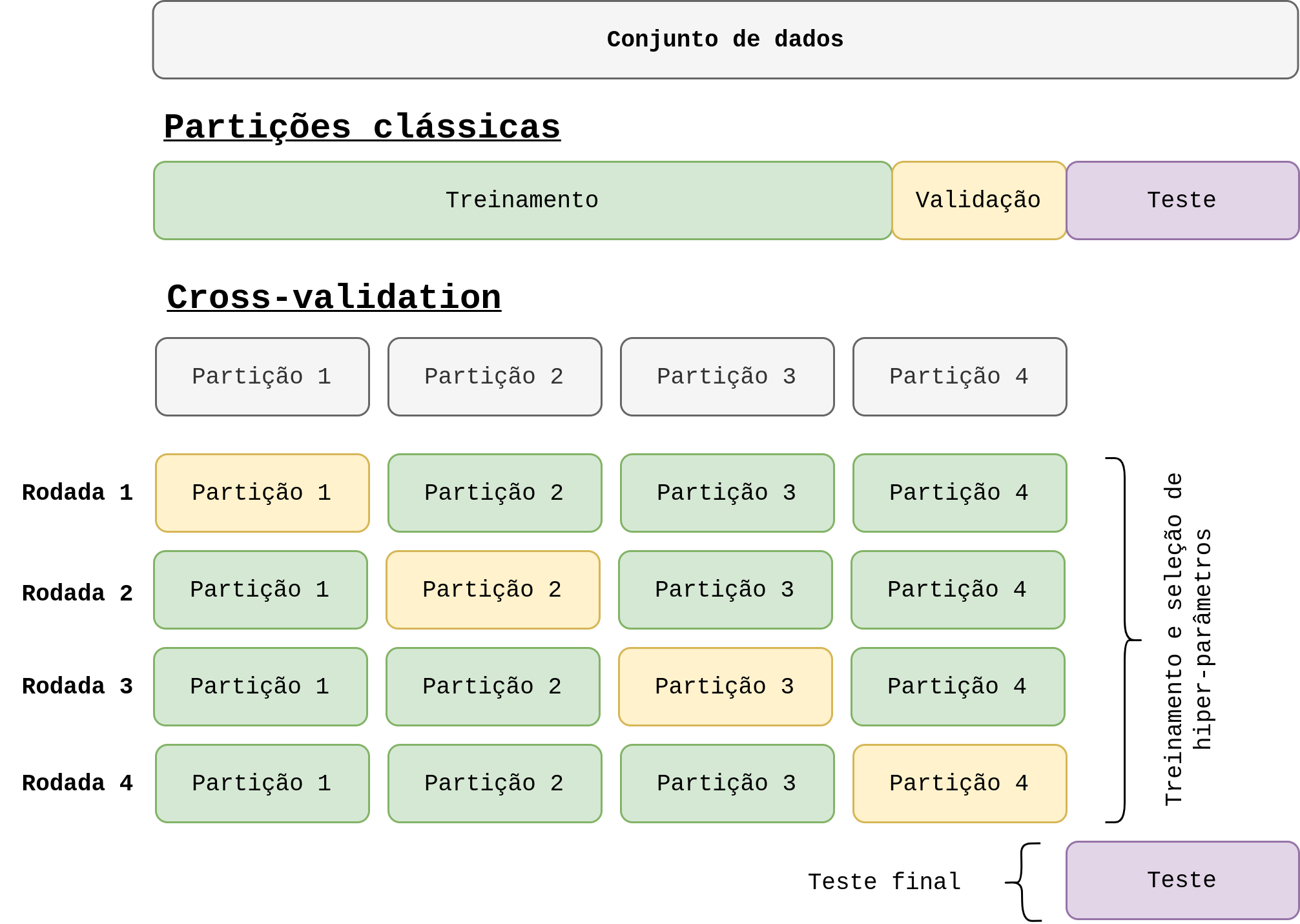
Utilizando cross-validation
A maneira mais utilizada e aceita para avaliar modelos treinados via dados é utilizando cross-validation (ou validação cruzada). Esse método é tão importante que a sklearn oferece várias maneiras de executá-lo. Neste post vamos explorar alguns deles, começando das formas mais automáticas até as que te dá mais controle e versatilidade. Todas as funções e métodos se econtram no subpacote sklearn.model_selection e eu nem preciso mais falar que para mais informações você deve consultar a documentação oficial.
Usando cross_val_score
A primeira maneira que vamos abordar é utilizando a função cross_val_score. Basicamente, essa função executa a validação cruzada de acordo com o número de folders (partições) informada via parâmetro cv. Na sequência ela retorna uma métrica de performance para cada folder de teste. Por padrão, essa métrica é a acurácia (para classificação).
from sklearn.model_selection import cross_val_score
acuracias = cross_val_score(knn_pipeline, X, y, cv=5)
print("acuracias:", acuracias)
print("acuracia final:", np.mean(acuracias), "+-", np.std(acuracias))
Podemos definir qual métrica a função deve calcular utilizando o parâmetro scoring:
f1s = cross_val_score(knn_pipeline, X, y, cv=5, scoring="f1_macro")
print("F1-macros:", f1s)
print("F1-macros:", np.mean(f1s), "+-", np.std(f1s))
Perceba que essa função faz todo processo de maneira automática e já retorna as métricas tirando a média de cada partição, neste exemplo, 5 partições.
Importante: para evitar vazamento de dados entre as partições, é importante você utilizar o Pipeline englobando o modelo e o transformador/normalizador. Já falamos sobre isso na Parte 1, mas nunca é demais relembrar. Para entender o motive, veja o post Avaliação de modelos, cross-validation e data leakage.
Usando cross_validate
Essa função é bem parecida com a anterior. A principal diferença é que ela permite definir várias métricas de retorno. Além disso, ela sempre retorna o tempo que os métodos fit() e score() levaram para ser executado:
from sklearn.model_selection import cross_validate
nome_metricas = ['accuracy', 'precision_macro', 'recall_macro']
metricas = cross_validate(knn_pipeline, X, y, cv=5, scoring=nome_metricas)
for met in metricas:
print(f"- {met}:")
print(f"-- {metricas[met]}")
print(f"-- {np.mean(metricas[met])} +- {np.std(metricas[met])}\n")
Como você pode observar, o método retorna um dicionário com a média de cada umas das métricas obtidas por meio das partições.
Gerando predições via cross_val_predict
Essa função permite obter as predições do modelo ao invés de apenas as métricas finais. Neste caso, cada predição será obtida para o conjunto de teste de cada uma das partições. Em outras palavras, se cv=5, por exemplo, o modelo vai ser treinado para 4 partições e testado em 1, que gera as predições. Ao final das 5 execuções, os resultados são concatenados e retornados.
from sklearn.model_selection import cross_val_predict
pred = cross_val_predict(knn_pipeline, X, y, cv=5)
print(pred)
Podemos utlizar o parâmetro method para escolher qual predição será retornada, nesse exemplo, vamos retornar a probabilidade de cada classe:
pred_prob = cross_val_predict(knn_pipeline, X, y, cv=5, method="predict_proba")
print(pred_prob[0])
Uma observação muito importante é que as predições retornadas para dos dois últimos exemplos podem ser diferentes pois a cada execução o modelo será retreinado para cada partição (que pode ser diferente). Obviamente isso não é muito desejável e vamos lidar com esse problema nas próximas sub-seções.
Obtendo mais controle utilizando KFold e StratifiedKFold
Diferentemente das funções mostradas até aqui, a KFold e a StratifiedKFold são classes que vão criar as cada uma das partições da validação cruzada para te dar mais controle da execução desse método de avaliação de modelos. Ambas retornam generators com os indices dos arrays de dados, de acordo com o número de partições escolhidas, que devem ser utilizados criar de fato cada uma das partições.
Usando a KFold
A ideia da KFold é simples: retornar os indices de cada partição de maneira aleatória. Esses indices são obtidos de acordo com o número de partições e são utilizados para construir-las a partir dos dados:
from sklearn.model_selection import KFold
n_folders = 5
cross_val = KFold(n_splits=n_folders, shuffle=True, random_state=32)
dados_cv = {f"folder_{f+1}": {"treino": None, "teste": None} for f in range(n_folders)}
k = 1
for indices_treino, indices_teste in cross_val.split(X):
dados_cv[f"folder_{k}"]["treino"] = (X[indices_treino], y[indices_treino])
dados_cv[f"folder_{k}"]["teste"] = (X[indices_teste], y[indices_teste])
k+=1
print(dados_cv.keys())
Neste exemplo, utilizamos n_splits = 5, que determina o número de partições. Além disso, embaralhamos os dados com um valor definido em random_state para garantir a reprodução dos experimentos. Definimos também um dicionário para colocarmos os dados de cada uma das partições. O método split() recebe os dados de treino X e retorna um generator na qual iteramos para obter os indices das partições. Na sequência, armazenamos os dados dentro do dicionário dadod_cv.
Usando a StratifiedKFold
A classe StratifiedKFold é uma variação da KFold na qual a principal diferença é que a mesma estratifica os dados de acordo com as classes do problema. Em outras palavras, ela balanceia a quantidade de amostras de cada classe entre as partições. Isso é relevante se o dataset é desbalanceado, algo comum em ML. O uso é basicamente o mesmo da KFold, porém, precisamos o método split() recebe tanto X como y, uma vez que ele necessita das classes para fazer o balanceamento:
from sklearn.model_selection import StratifiedKFold
n_folders = 5
cross_val_strat = StratifiedKFold(n_splits=n_folders, shuffle=True, random_state=32)
dados_cv_strat = {f"folder_{f+1}": {"treino": None, "teste": None} for f in range(n_folders)}
k = 1
for indices_treino, indices_teste in cross_val_strat.split(X, y):
dados_cv[f"folder_{k}"]["treino"] = (X[indices_treino], y[indices_treino])
dados_cv[f"folder_{k}"]["teste"] = (X[indices_teste], y[indices_teste])
k+=1
print(dados_cv.keys())
Usando o generator como entrada de outras funções
Uma utilidade interessante para a instâncias obtidas para as classes KFolde StratifiedKFold (que nos exemplos anteriores denominamos cross_val e cross_val_strat) é que elas podem ser utilizadas pelas funções cross_val_predict, cross_validate e cross_val_predict. Basicamente, o parâmetro cv, comum às três funções, pode receber seja um inteiro ou uma instância das classes já mencionadas. Isso garante que as partições sejam exatamente as mesmas e evita o problema citado quando usamos method="predict_proba":
pred = cross_val_predict(knn_pipeline, X, y, cv=cross_val)
pred_prob = cross_val_predict(knn_pipeline, X, y, cv=cross_val, method="predict_proba")
print(pred[112])
print(pred_prob[112])
Além disso, também podemos tomar controle do loop de execução da validação cruzada do modelo. Iniciamos esse post executando o modelo N vezes para partições diferentes de treino e teste. Podemos fazer o mesmo, mas agora usando as partições salvas em data_cv. Isso é basicamente o que as funções anteriores fazem por trás dos panos. Mas por algum motivo, você pode querer controlar esse loop:
acuracias = list()
for folder in dados_cv:
knn_pipeline.fit(dados_cv[folder]["treino"][0], dados_cv[folder]["treino"][1])
ac_i = knn_pipeline.score(dados_cv[folder]["teste"][0], dados_cv[folder]["teste"][1])
acuracias.append(ac_i)
print(f"{folder}: {ac_i}")
print("\n- Acurácia das folders:")
print(f"Media: {round(np.mean(acuracias) * 100, 2)}%")
print(f"Desvio padrão: {round(np.std(acuracias) * 100, 2)}%")
Considerações finais
Neste post discutimos duas maneiras de avaliarmos os nossos modelos utilizando funções, métodos e classes da sklearn. O foco maior foi na validação cruzada, dado sua relevância e importância. É importante levarmos em consideração que o problema de classificação que estamos lidando é bem simples e serve apenas para aprendizado. Além disso, existem outras formas de se obter a validação cruzada via sklearn. Apresetamos apenas as principais, mas você pode facilmente utilizar outras checando a documentação desse módulo em específico. Para concluir, todos os trechos de códigos aqui utilizados foram feitos com KNN. Mas como já sabemos, tudo funciona perfeitamente com outros estimadores, graças a excelente padronização da biblioteca. Fica como atividade, você modificar tanto o estimador quanto o problema no Pipeline utilizando o nootebook no nosso repositório do Github.
Para o próximo post, vamos abordar um outro problema, com mais estimadores e outras maneiras de verificar o desempenho dos modelos.


Deixe um comentário